KISTI, 슈퍼컴퓨터 활용 코로나19 감염 저항성 연구 발표
최나은 2022-07-13 View. 19,979첨부파일(2)
KISTI, 슈퍼컴퓨터 활용 코로나19 감염 저항성 연구 발표
- 병렬컴퓨팅 기술로 유전적 다양성에 따른 바이러스 침투 기작 밝혀 -
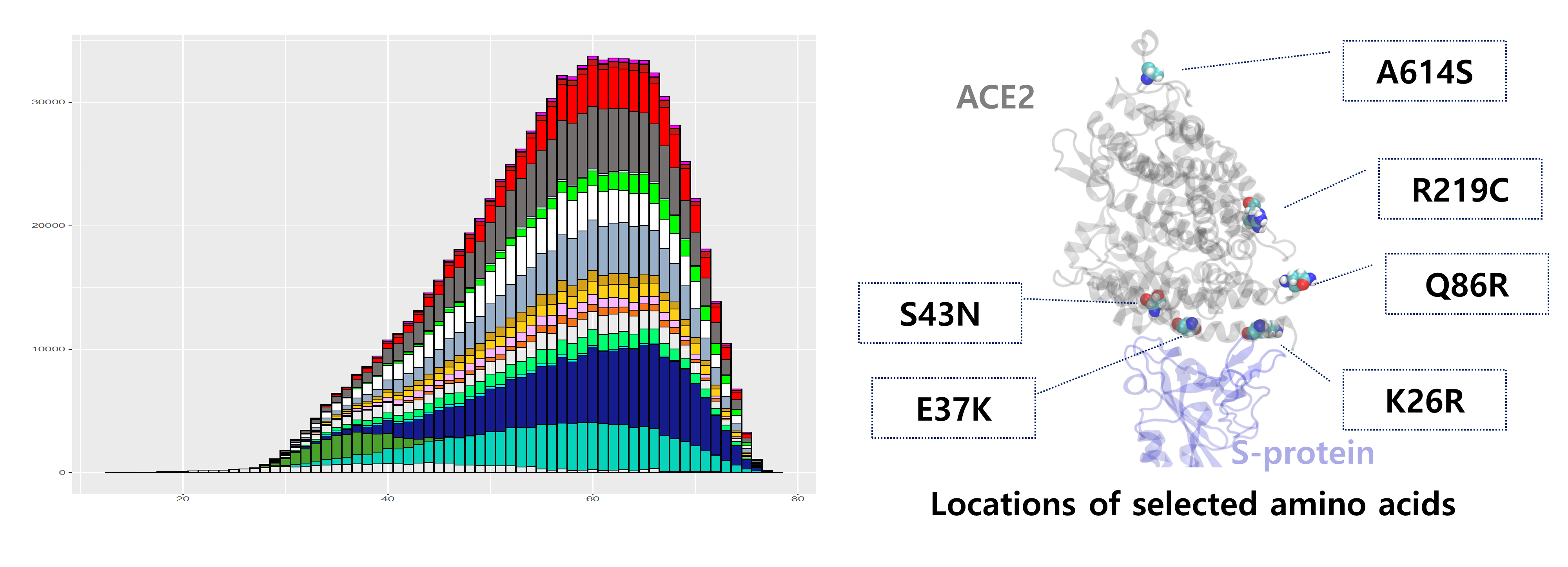
<20만 명 규모의 바이오빅데이터 분석과 슈퍼컴퓨터 기반의 SARS-CoV-2 구조 분석
(좌) 20만 명 규모의 의료기록 패턴과 유전변이 통합 분석 그래프
(우) 슈퍼컴퓨터 기반의 SARS-CoV-2 구조 분석 가시화 결과>
한국과학기술정보연구원(원장 김재수, 이하 KISTI)이 KISTI 슈퍼컴퓨터* 누리온을 활용해 개인 유전형 변이에 따른 hACE2 단백질 구조의 다양성을 탐색하고 이에 따른 COVID-19 바이러스 SARS-CoV-2와 결합에너지의 변화를 도출했다고 밝혔다.
*슈퍼컴퓨터란 www.top500.org에 공개된 계산 성능 전 세계 기준 500위 안의 컴퓨터를 뜻함. 통상 수천대 이상의 계산 서버가 연결돼 있음.
hACE2 단백질은 코로나 바이러스가 인간 세포에 접합하기 위한 세포막의 단백질이며, SARS-CoV-2와의 결합에너지 크기가 감염 저항성에 영향을 미칠 것으로 기대되고 있다.
본 슈퍼컴퓨팅 기반 연구는 영국 UKBiobank에서 수집된 20만 명 규모의 유전변이 및 COVID-19 감염 검사 데이터를 분석하고, 해당 분석결과와 KISTI 슈퍼컴퓨터 누리온을 활용해 계산한 SARS-CoV-2 바이러스 결합에너지를 비교하여 수행했다.
특히 한국과학기술정보연구원 슈퍼컴퓨팅응용센터 바이오의료팀은 생물정보학 분야의 전문성을 바탕으로 영국의 대규모 유전변이 데이터를 분석하고, 슈퍼컴퓨팅 계산으로 SARS-CoV-2의 스파이크 단백질과 hACE2 단백질의 결합력 측정을 빠르고 효과적으로 수행했다.
연구책임자인 백효정, 서상재 박사는 “이번 hACE2 관련 유전변이에 따른 COVID-19 바이러스 결합 연구는 영국의 실제 COVID-19 감염 검사 데이터를 활용해 그 의미가 크다.”며 “국내에서도 바이오 빅데이터가 축적되고 이를 통해 신규 감염병 대응 연구가 효율화되기를 기대한다.” 라고 말했다.
본 연구 내용은 생물 정보학 분야의 주요 국제 저널인 ‘PLoS Computational Biology’에 온라인 게재**되었다.
**Paik et al, Analysis of the docking property of host variants of hACE2 for SARS-CoV-2 in a large cohort (2022년 7월 11일 온라인 게재, PLoS Computational Biology).
KISTI 정민중 슈퍼컴퓨팅응용센터장은 “팬데믹 이후 유전체 및 보건 의료 분야의 슈퍼컴퓨팅 응용연구가 활발해져 다양한 사회문제가 해결되기를 기대한다.”라고 밝혔다.

본 저작물은 “공공누리 제4유형(출처표시, 상업적 이용금지, 변경금지)” 조건에 따라 누구나 이용할 수 있습니다.
- 담당부서
- 담당자박성욱
- 연락처042-869-1610

 Delete Article!
Delete Article!